단백질은 구조-기능적-진화적으로 연관된 패밀리들로 분류할 수 있습니다.
단백질 패밀리는 1000~2000여 종이 있다고 합니다.
EGFR 단백질 및 그 패밀리에 대해 구조를 비교해보겠습니다.
EGFR은 교과서에도 잘 나와있을 정도로 유명하고, 많은 선행 연구 결과들이 있고, 관련 약물도 많이 개발되어있는 단백질이라서 신약개발을 공부할 때 좋은 타겟입니다. 또한 암에 대한 타겟 단백질이고 변이와 관련이 있기에 정밀 의료의 기본 개념을 이해하기에도 좋습니다.

EGFR(Epidermal growth factor receptor, 표피생장인자수용체, HER1)는 수용체 역할을 하는 막 단백질이자, tyrosine kinase입니다. 이 단백질은 구조적으로 여러 영역(domain)으로 나뉘어 있는데, 그중 receptor 영역은 세포 밖에 위치하고, kinase 영역은 세포 안에 위치합니다. receptor 영역은 세포 외부에서 메신저 (EGF 및 유사체)와 결합하면 kinase 영역에 구조 변화가 일어나는데, 이렇게 구조 변화가 일어나면 kinase로서의 기능이 활성화됩니다.
EGFR이 인산화시키는 대상은 다른 EGFR 혹은 EGFR과 같은 패밀리(HER)에 속하는 단백질들입니다. (HER2, HER3, HER4)
EGFR은 EGFR 혹은 다른 HER 패밀리의 단백질과 dimer를 이루고, 서로를 인산화합니다.
그리고 이렇게 인산화가 되면 신호전달 경로상에 있는 다른 단백질에 신호가 전달되고, 최종적으로 핵 안까지 전달되어 세포 성장과 세포분열을 일으키는 특정 유전자의 전사를 조절합니다.
암은 세포가 과도한 분열을 일으키는 병으로, EGFR 같은 성장인자가 암에서 중요한 기능을 하는 경우가 있습니다. 특히 EGFR의 L858R 변이가 일어날 경우, EGF 같은 메신저가 결합하지 않아도 kinase로서의 기능이 활성화되어버려 과도한 신호를 전달하고 세포 분열을 일으킵니다. 이 변이가 일어날 경우, 단백질의 포켓 구조가 크게 변합니다. 이 때문에 gefitinib에 대한 결합력이 크게 상승해서, 정상 EGFR에 비해, 암세포의 EGFR에 대한 선택성이 좋아집니다. 단백 비소세포성 폐암의 주요 원인이자 타겟으로 알려져 있습니다. 생체는 일반적으로 특정 단백질, 혹은 신호에 문제가 생겨도 바로 병으로 이어지지 않도록 우회경로나 조절 장치가 존재하여 높은 안정성을 가지고 있는데, 반면에 이미 질병이 발생했다면, 복합적인 원인에 의한 것일 가능성도 생각할 수 있습니다. 몇 가지 기능이 함께 고장 났기에, 그것을 해결하는 것이 쉽지 않은 일이 됩니다. 그런데 EGFR L858R 변이의 경우는 단일 변이만으로도 암을 유발할 수 있고, 반대로 해당 변이에 의한 암임을 식별할 경우는 현재 개발된 약물의 치료 효과가 높다고 합니다.
EGFR과 같은 tyrosine protein kinase에 속하는 막단백질들이 여럿 있는데, DUD-E 데이터에 있는
EGFR, TGFR1, VGFR2 를 비교해보겠습니다. 이들은 같은 슈퍼 패밀리에 속합니다.
서열 및 단백질에 대한 정보는 uniprot에서 찾아볼 수 있습니다. 일단 인간 단백질을 선택합니다.
그리고 pdb에서 kinase domain에 대한 단백질 구조를 얻습니다. DUD-E 데이터에서 사용한 pdb id를 사용하겠습니다.
EGFR:
https://www.uniprot.org/uniprot/P00533
https://www.rcsb.org/structure/2RGP
EGFR canonical sequence:
https://www.uniprot.org/uniprot/P00533.fasta
TGFR1:
https://www.uniprot.org/uniprot/P35968
https://www.rcsb.org/structure/3HMM
VGFR2:
https://www.uniprot.org/uniprot/P35968
https://www.rcsb.org/structure/2P2I
이 세 단백질 사이의 서열 유사성은 별로 높지 않습니다. 도메인을 나누지 않고 nwalign을 돌려보면,
(https://zhanggroup.org/NW-align/)
TGFR1-EGFR Sequence identity: 0.126
VGFR2-EGFR Sequence identity: 0.244
VGFR2-TGFR1 Sequence identity: 0.314
입니다. 도메인을 나눈 후에 계산하는 편이 더 정확할 것이지만, 여기선 단지 서열 유사성이 크게 높지 않다는 것을 보여주기 위한 것이라 별로 정확도는 신경 쓰지 않았습니다.
kinase domain에 대한 단백질 구조 유사성은
TMalign 된 TMscore 로 (https://zhanggroup.org/TM-align/)
TGFR1-EGFR : 0.75935 (뒤의 단백질을 기준으로 normalize 되었습니다.)
VGFR2-EGFR : 0.79511
VGFR2-TGFR1 : 0.74911
TMscore에서 0.4 이상이면 눈으로 봤을 때 정렬이 잘 되고 유사하게 보이기 시작하는 정도입니다.
정렬된 그림을 보여드리겠습니다.

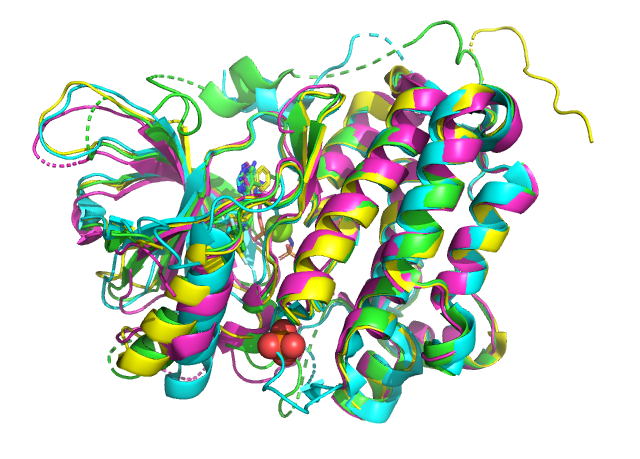
색깔별로 EGFR (2RGP), TGFR1 (3HMM), VGFR2 (2P2I) 입니다.
위 그림에서 kinase 도메인의 구조가 서로 유사함을 볼 수 있습니다.
kinase에서 주로 약물이 결합하는 위치는 ATP binding pocket입니다.(이외에 allosteric도 있긴 하지만...)
만약, ATP binding pocket의 구조가 유사하다면 약물에 대한 결합력도 유사할 가능성이 높습니다.
이는, multi kinase inhibitor가 존재할 가능성이 높다는 의미이지만, 반대로 selectivity를 주기는 어렵다는 의미입니다.
그러면 이 세 단백질의 포켓 유사성은 어떨까요? 포켓 유사성도 측정하는 메서드가 있긴 하지만, 제가 사용하는 것은 없기에 그냥 이미지만 보여드리겠습니다.
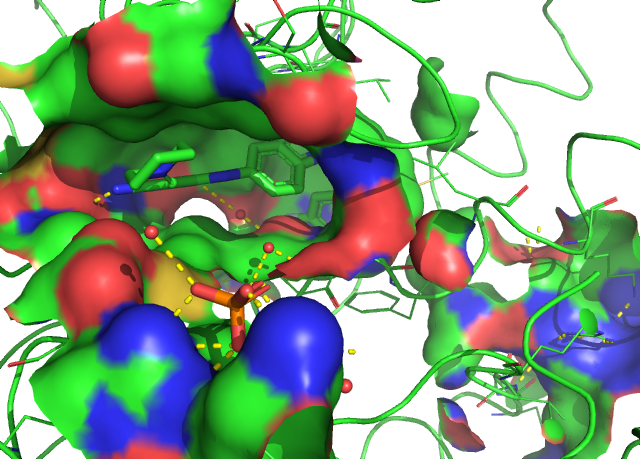
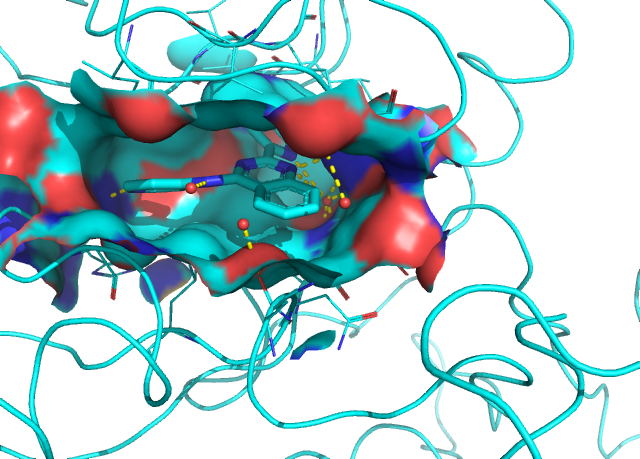
EGFR (2RGP), TGFR1 (3HMM) 의 포켓 구조는 유사합니다. 둘 다 넙적하게 생긴 분자들이 붙기 쉽게 생겼습니다.
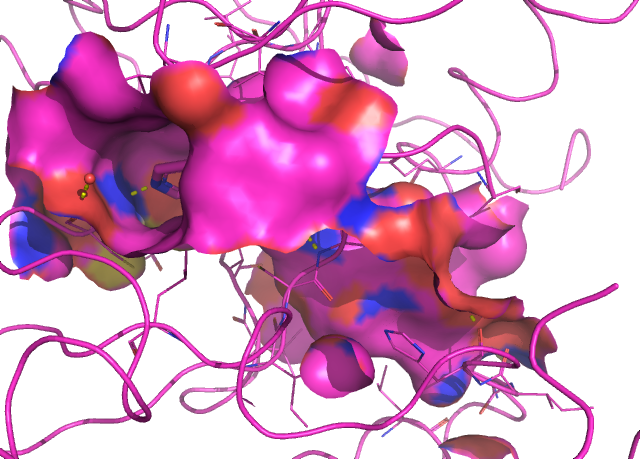
그런데, VGFR2 (2P2I) 의 포켓 구조는 터널 형태로(반대편이 뚫려있습니다.) 길쭉하게 생긴 분자가 터널을 채우고 있습니다.
포켓 구조만 봐도 EGFR과 VGFR2에 동시에 결합하는 약물을 설계하는 것은 상당히 어려운 일임을 알 수 있습니다.
DrugBank의 데이터에서도 양쪽을 다 타겟하는 약물은 거의 없습니다. 하나 본것이 있긴 한데, 그건 타겟 kinase가 수십 개라서 실제로 결합력이 있는지 좀 의문이 들더군요.
그럼 다음은 EGFR과 같은 HER 패밀리의 HER2, HER3, HER4의 서열과 구조를 비교해보도록 하겠습니다.
HER2:
https://www.uniprot.org/uniprot/P04626
HER3:
https://www.uniprot.org/uniprot/P21860
HER4:
https://www.uniprot.org/uniprot/Q15303
HER2 HER1 Sequence identity: 0.520
HER3 HER1 Sequence identity: 0.446
HER4 HER1 Sequence identity: 0.539
슈퍼 패밀리의 sequence identity 보다 패밀리 사이의 sequence identity가 더 높음을 볼 수 있습니다.
색깔별로 HER1 (2RGP), HER2 (3PP0), HER3 (3LMG), HER4 (3BBT) 입니다.
TMalign에서 TMscore는
HER2 HER1: 0.88354
HER3 HER1: 0.87945
HER4 HER1: 0.89580
입니다. 같은 패밀리에 속한 단백질끼리 구조 유사성이 상당히 높음을 볼 수 있습니다.
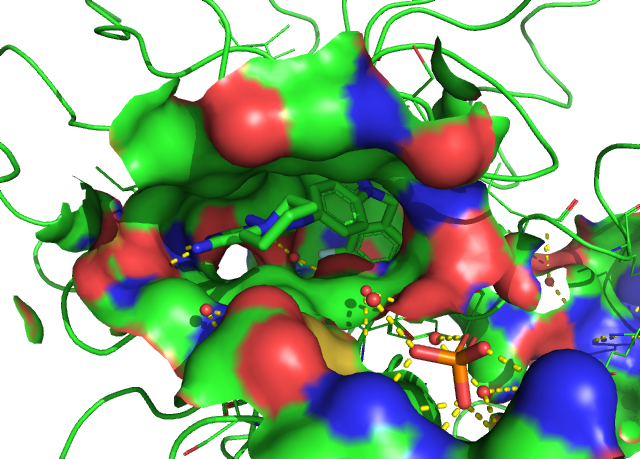
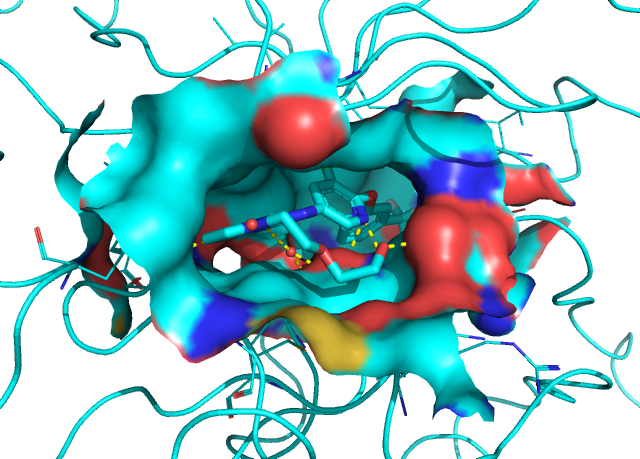


포켓의 구조도 서로 유사합니다. HER3의 경우만 결합된 Ligand가 ATP라서 포켓 구조를 비교하기 조금 좋지 않네요.
확인은 안 해봤지만, ChEMBL 등에서 약물 결합력 실험 데이터를 가지고 비교하면 서로에 대한 코릴레이션이 높게 나올 것을 예측할 수 있습니다.
만약 Template based modeling을 한다면, 가장 적절한 Template은 같은 패밀리에 속하는 단백질입니다.
HER4의 구조를 모른다면 HER1, HER2, HER3 구조를 템플릿으로 사용하면 거의 정확히 예측할 수 있을 것입니다.
그런데 슈퍼 패밀리만 돼도 위험성이 생깁니다. 만약 VGFR2를 사용해서 EGFR의 구조를 예측한다면, 전체 구조는 유사하게 나오겠지만, 포켓 부근의 구조를 얼마나 정확히 예측할 수 있을지가 문제입니다.
단백질 구조 예측을 단백질 구조 기반 신약개발에 활용하기 위해선 바인딩 포켓에 대한 구조를 잘 맞추고, 약물 결합력 시뮬레이션에 적합한 구조를 얻어야 합니다.
이번 글은 이것으로 마무리하고 다음에는 EGFR의 뮤테이션에 따른 구조 차이와 약물 결합력에 대한 글을 포스팅하겠습니다.
'Drug' 카테고리의 다른 글
| 타겟 단백질의 종류와 약물 (0) | 2021.10.23 |
|---|---|
| 도서 추천: 의약화학 (0) | 2021.09.24 |
| 의약화학: ACE 억제제 (고혈압 치료제) (2) | 2021.09.24 |
| 신약 개발 과정과 약물 재설계 (0) | 2021.09.09 |
| 의약화학: 항궤양제 H2 길항제 (0) | 2021.07.25 |