저에게 가장 의미 있는 두 편의 논문중 하나입니다.
저는 좋은 연구를 하고 싶은 분들께 이 연구를 추천해드리고 싶습니다.
당시에 좋은 연구란 무엇인가에 대해 고민을 많이 했습니다. 그리고 이 논문이 제 결론이었습니다.
좋은 아이디어는 그것만으로도 연구로서의 가치를 가지고 있습니다.
다만 논문은 개판으로 못썼고, 논문의 구성도 형편없습니다.
논문을 작성하는 분들에게는 구성이 안 좋은 논문의 예시로서 보여드릴 수 있겠습니다.
이 논문에서 단백질 도메인 경계 예측문제를 예측하기 위한 당시로선 획기적인 방법을 제시하였습니다.
제목은 Condo: protein domain boundary prediction using coevolutionary information
입니다.
2018년도에 나온 논문이고, 에디터는 Alfonso Valencia 입니다.
https://academic.oup.com/bioinformatics/article/35/14/2411/5221017
https://academic.oup.com/bioinformatics/article/35/14/2411/5221017
academic.oup.com
단백질 도메인에 대해서 먼저 설명드리겠습니다.

Fig. S1의 (A) 는 단백질의 3D 구조를 cartoon으로 표현한 것입니다. 빨간색과 파란색은 각각의 도메인을 표시합니다. 단백질은 아미노산 염기 서열의 체인으로 이루어져 있는데, 3D 공간상에서 접히면서 고유의 구조를 가집니다. "단백질 도메인이란, 안정되고 컴팩트한 구조인데, 이는 구조의 단위이자, 기능의 단위이고, 진화의 단위입니다." 사실 이 말 한마디에 이 연구의 모든 아이디어가 담겨있습니다.
문제 안에 답이 있다. 라는 말이 있습니다. 이 연구가 바로 그런 경우입니다. 이 연구는 단백질의 도메인에 대한 정의를 재해석함으로써 시작됩니다.
단백질 도메인 경계 예측이라는 문제는, 단백질의 서열로부터, 도메인의 경계를 예측하는 문제입니다. 이 문제가 중요했던 이유는 당시 단백질 구조예측이나 단백질 구조 결정 실험은 단백질 도메인 단위에서 진행했기 때문입니다. 도메인은 folding의 단위이기에 도메인 경계에서 단백질 체인을 잘라도 각각의 구조는 유지됩니다. 단백질의 서열이 길어지면 계산량은 서열에 비례하는 것이 아니라, 대충 말해서 서열의 제곱에 비례합니다.(뭐 방법론마다 조금씩 차이는 있습니다만...)
구조 결정 실험에서도 크기가 클수록 실험이 어려워집니다. 예를들어 EGFR 같은 RTK (리셉터 티록신 카이네이즈) 들은, 세포 밖의 receptor 역할을 하는 도메인과, 세포 내의 kinase 역할을 하는 도메인이 있는데, 보통 PDB에서 구조들을 보면 이중 한 도메인에 대한 결정 구조밖에 없습니다. 도메인 단위에선 어느 정도 견고하지만, 도메인 간의 움직임은 유동적이고, 특히 그 사이에는 세포막이 있으니 전체 단백질에 대해서 한 번에 실험으로 구조를 찾는 것은 매우 어렵습니다.
단백질 도메인은 구조, 기능, 진화의 단위이기에 각각의 분석도 도메인 단위에서 진행해야 하는 면도 있습니다.
Fig. S1의 (B) 에서 Query 라 쓰여있는 줄이 이 단백질의 아미노산 서열입니다. 서열의 길이는 283이고, 100 단위로 줄을 바꿔서 적었습니다.
단백질 도메인 경계 예측에서 가장 강력한 방법은 단백질 구조 예측에서와 마찬가지로 template based method (TBM)입니다. 서열이 비슷하면 구조가 유사할테니, query 단백질의 서열과 비슷한 단백질을 찾아서 서열을 정렬하고, 그 단백질의 도메인 정보를 참고하는 것입니다. 당연히 구조가 알려진 homology (상동) 단백질이 없다면 사용할 수 없는 방법입니다.
그래서 단백질 도메인 경계 예측 문제는 TBM과 sequence based method로 나뉩니다. 당연히 전자가 성능은 높지만, 전자로는 풀 수 없는 문제가 있기에 후자가 필요합니다. 이 논문은 후자에 대한 방법론입니다.
사실 이 논문이 나오기 이전에 도메인 경계 예측 성능은 처참했습니다. 이전 연구들은 주로 경계 주변의 20개 정도의 아미노산 서열과 sequence profile (Position specific scoring matrix, PSSM), 이차구조 정보, solvent accessibility 같은 local feature 만을 사용하였습니다. 단백질 도메인 경계를 결정하는 요소가 전부 들어가야 도메인 경계를 예측할 텐데... local 서열에 의해서 도메인 경계가 결정되지 않으니 long range feature가 필요하지만, 그것을 활용한 사례가 없었습니다.
그런데 아마도 아시는분들은 다 아시겠지만, coevolutionary information 은 단백질의 residue-residue contact과 밀접한 관련이 있고, 이것을 이용해서 단백질 구조예측 정확도가 환상적으로 향상되었습니다. 사실 이 연구의 아이디어도 contact based modeling을 연구하던 중에 단백질 3D 구조와 contact prediction 결과를 비교하다가 떠오른 것입니다. coevolution이나 contact은 long range feature라고 할 수 있습니다.
서로 동일한 domain 에 속한 residue 사이에선 contact 이 많이 있을 것이고 반면에 서로 다른 domain 에 속한 residue 사이에는 contact이 적을 것입니다. contact이라는 표현을 공진화로 바꿔보죠. domain 이 진화의 단위라면, 공진화는 domain 내에서 활발히 일어나지만, 서로 다른 domain 내에선 공진화가 적을 것입니다.
일단 domain이 진화의 단위라는것을 먼저 확실히 볼 수 있는 것이 Fig. S1 (B)의 Temp2,3,4,5입니다.
이들은 Uniref DB의 sequence들 중에서 query에 대한 homology 들을 탐색해서 정렬한 것들인데, 신기하게도 몇몇 서열들은 한 도메인까지만 정렬이 됩니다. temp2,3은 N-terminal domain(파란색) 에만 정렬되고, temp4,5는 C-terminal domain(빨간색) 에만 정렬됩니다.
왜그러냐면... 도메인이 진화의 단위이기 때문입니다. 여기에 해당되는 사례가 EGFR과 KIT 이라는 두 단백질입니다. 둘 다 RTK입니다. 이 두 단백질의 kinase domain 구조는 서로 유사합니다.
그런데 신기하게도 receptor domain은 전혀 다르게 생겼습니다. KIT의 receptor domain는 오히려 면역 단백질들의 구조와 더 닮았습니다. 즉, kinase domain끼리는 서로 (super) family이지만, receptor domain 은 그렇지 않습니다. 따라서 homology 서열들을 찾아서 정렬하면, domain 단위에서 정렬이 됩니다.

이런 부분적으로 정렬된 서열들을 평균해서 그린것이 Fig S2 (A)입니다. 부분적으로 정렬된 서열들을 비주얼라이즈 한 것인데, 수식 설명은 생략하겠습니다. 사실 이것만 보여줘도 그냥 도메인 경계가 어디인지 빤히 보이죠. 그래도 머신에 입력으로 넣기 위해서 좀 가공을 했습니다만...
그런데, 이렇게 부분적으로만 도메인 단위에서 정렬된 서열만 있는것이 아니라, Fig S1(B)의 temp1처럼 query와 서열 전체가 정렬된 서열들도 있습니다. 경우에 따라서 부분적 정렬 정보가 거의 없는 경우도 있습니다.

Fig. S3 (A) 가 그런 경우입니다. 이것만 봐서는 도메인 경계에 대한 정보가 전혀 보이지 않죠.
그럼 여기서 coevolutionary information을 확인해봅시다. (C)는 DCA (direct coupling analysis) 중 하나인 CCMpred 결과입니다. 도메인 내의 residue 사이의 공진화가 도메인 간의 residue의 공진화보다 훨씬 활발한 것을 볼 수 있습니다.
사람이 눈으로 봐도 도메인 경계가 어디인지 보이죠. 이 그림만 가지고도 공진화로 도메인 경계를 예측하겠다고 하는 아이디어가 얼마나 타당한지 알 수 있습니다. 뭐.. 그래도 딱 하나 보여주고 그럴 순 없고, 통계적으로 유의미해야 하긴 하지만...
(D) 각 residue 를 기준으로 cluster를 나눴을 때의 modularity 값들을 계산한 것입니다. domain 경계 근처 modularity가 peak 인 것을 볼 수 있습니다.

Fig. S4 는 modularity peak 만 이용해서 도메인 경계를 뽑았을 때, single-domain protein과 multi-domain protein이 얼마나 잘 분류되는가를 확인한 것입니다. 이전 메서드들에 비하면 이것만 가지고도 충분히 유용하다는 것을 보여줍니다.
아이디어 차원에서는 이전 연구들과 비교해서 차별성이 있고, 될 수밖에 없는 연구라는 것을 보였습니다.
이걸로 연구가 끝나면 참 좋겠지만... 연구는 그렇게 깨끗한 일이 아니죠. 디테일은 참 더럽습니다. 그리고 처음 하다보니 좋지 않은 옵션도 많이 선택했습니다. 원래 첫 논문은 아이디어 제시하는 것이 큰 일이고, 그것을 최적화하는 것은 후속 연구자들의 몫이죠.. (이 논문이 나온 후에 Yang Zhang 그룹에서 아쉬운 부분들을 잘 해결해서 좋은 논문을 썼더군요.)
위의 분석들이나 피처들은 단백질의 도메인이 1개이거나 2개일 경우에 적절하지만,3개이상이라면 좀 적절하지 않은 면이 있습니다. 어쨌건 예측은 가능하도록 위의 정보들을 feature로 넣고 ML 모델을 만들었습니다.
각 residure 별로 도메인 경계인가를 예측하는 모델이었습니다. 도메인 경계가 2개 이상이 될 수도 있어서 regression으로 다룰 수 없고 classification으로 해야 합니다. 이중에 예측 값이 얼마 이상이 나오는 곳들을 경계 후보로 선택하도록 하였습니다. 특정 residue 근처에서 경계 후보가 여러개가 나오면 그중에 제일 적절한 것을 선택해야 하는데, 예측값의 peak 이 서로 30 residue 정도 떨어져 있으면 난감합니다... 둘 다 도메인 경계인지, 하나만 경계인지... 이런 다양한 어려운 문제들이 있었습니다.
ML은 이때 처음 써보다보니 아쉬운 것도 참 많았습니다.
그리고 dis-continuous domain이라는 골치 아픈 것이 있었는데, 이 논문에선 거기까지 다루진 못했습니다. 그것도 사실 DCA map을 보면 알 수 있긴 하지만...
그리고 이 논문 구성이 매우 안좋다고 생각하는 이유인데, 이 위에 있는 Fig. 들에 죄다 S가 붙습니다.
좋은 그림들을 Supplementary Data 에다 넣어버리고 논문 메인은 시시한 그림들만 넣어버렸네요.
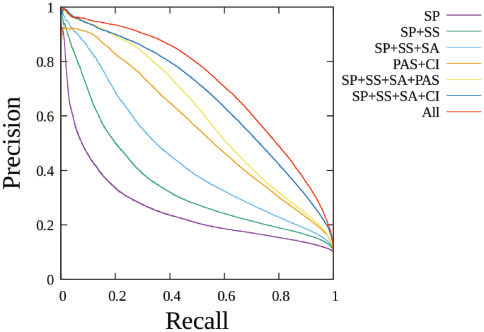
최종 예측 대상은 도메인 경계가 어디에 있는가? 이지만, 머신에 그것을 직접 학습시키진 못했고,
각 residue 에 대해서 도메인 경계에서 5, 10, 15, 20 residue 이내에 있는가? 를 예측시켰습니다. (Fig 3.)
피처에 대한 설명입니다.
이 3개는 short range information입니다.
Sequence profiles (SPs) such as position-specific scoring matrices
predicted secondary structure (SS),
predicted solvent accessibility (SA)
이 둘은 long range information 입니다.
coevolutionary information (CI), partially aligned sequences (PASs),
이 그림을 보면 이전의 short range feature만을 사용한 방법들의 한계가 여실히 보이고, long range interaction에 대한 정보가 얼마나 중요한지 알 수 있습니다.
이거 외에도 논문에 몇가지 더 지저분한 것들이 있지만... 리프로듀스 할게 아니라면 굳이 볼 필요는 없어서 생략합니다.
저는 이 연구에서 몇가지 배운 것이 있습니다.
연구에서 제일 중요한 것은 아마도 연구 주제를 정하는 것입니다.
아마도 대부분 대학원생들이 무엇을 연구해야 할지 고민하고, 좋은 연구주제를 정하지 못해서 고생하고 있을 것입니다.
뭐 저도 좀 오랜 시간 그랬습니다. 그럴 때는 큰 목표를 생각해보는 것입니다.
여기선 단백질 구조예측이 큰 목표가 되겠죠. 거기에 연관된 세부 주제들을 찾아보면, 작은 일들은 매우 많습니다. 물론 그 일들이 쉬운 일일지, 어려운 일일지는 별개이지만... 그래도 가치 있는 일들이 될 순 있습니다.
신약개발 같은 경우는 신약개발에 필요한 모든 세부 메소드들이 전부 좋은 연구 주제들입니다.
좋은 주제라고 해서 내가 해결할 수 없다면 좋은 연구가 될 수 없겠죠.
그럴 땐 흐름을 생각해보는 것이 좋습니다. 보통 연구들은 한 계단 한 계단씩 발전해갑니다.
그래서 역사를 공부하는 것처럼 이전의 연구들의 흐름을 보다보면, 흐름이 보이고, 그다음에 해야 할 주제가 보입니다.
이차구조 예측을 예로 들면 (남궁석 교수님께서 칼럼을 정말 훌륭하게 잘 쓰셨지만...) 매우 좁은 윈도우 사이즈에 대한 amino acid에 대한 통계 분석에서부터 AI 도입, sequence profile의 도입, CNN 도입 같은 것들이 순차적으로 도입되면서 성능이 개선되어갑니다. (딥마인드처럼 5계단 한번에 오르는 괴물들은 정말 예외입니다. )
잘 될 연구는 아이디어만 들어도 잘 될 것 같다는 생각이 들고, 쉽게 개념 증명을 할 수 있습니다.
그리고 결과도 잘 나오고, 동기와 흐름이 명확하기에 논문을 작성하기도 어렵지 않습니다.
제가 대학원생 시절부터 7년정도 시행착오를 해보면서 결국 알게 된 게, 좋은 연구가 오히려 쉽다는 것입니다.
좋은 연구는 대체로 아이디어가 명료하고, 그러기에 어떻게 보여야할지도 명쾌하게 진행됩니다.
물론 기술이나 데이터가 부족해서 할 수 없는 경우는 있습니다만...
내가 뭘 해야하는지 뭘 보여야 하는지 무슨 결과가 나와야 의미 있는지...
그런거 모르고 어떻게 좋은 연구가 되겠습니까?
그걸 보는 눈을 기르는 시기가 대학원 과정이고, 그걸 할 수 있게 되었을 때 졸업하는 것입니다.
사실 이 논문에는 뒷 사정이 좀 있습니다.
글을 너무 못쓰고 오타도 심하게 많아서 리뷰어들이 죄다 리젝 하라 했는데,
에디터 Alfonso Valencia 께서 메이저 리비전을 주셨습니다.
알고보니 이분이 단백질 coevolution에 대해서 이전부터 연구하고 계셨다고 하더군요.
누군지도 모르고 에디터로 골랐다는...
아무튼 이 글을 보고 고민하는 분들이 길을 찾을 수 있게 된다면 좋겠습니다.
저도 몇몇 분의 도움이 없었다면 졸업 못했을 것 같습니다.
그분들께 감사하며 저도 누군가에게 도움이 되고 싶습니다.
'Physics and Informatics' 카테고리의 다른 글
| Protein bioinformatics: 구조-기능-진화와 머신 러닝 (0) | 2023.02.09 |
|---|---|
| 아미노산 종류에 따른 residue-residue contact 비율 (0) | 2022.12.12 |
| 데이터 분석: 유사성과 정렬 (0) | 2022.08.05 |
| 인공지능은 물리학를 학습할 수 있을까? (0) | 2022.08.03 |
| 연구자로서 미래를 보는 것에 대해서 (0) | 2022.04.27 |