이전 글
https://novelism.tistory.com/259
https://novelism.tistory.com/260
설치는 이전에 했고...
일단 pdb파일에서 receptor와 ligand를 분리해야 합니다.
3HMM.pdb 를 3HMM_receptor.pdb 로 복사합니다.
편집은 메모장이나 워드패드를 사용하면 됩니다.
여기서 HETATM 중 residue id가 855인 행들을 선택해서 잘라냅니다.
이 855는 pdb 에서 사용하는 ligand 의 고유 ID가 됩니다.
잘라내고 남은 빈 줄은 지웁니다.
3HMM_855.pdb 라는 빈 파일을 만들고 잘라 넣은 내용을 붙여 넣습니다.
텍스트 파일을 만들어서 확장자면 pdb로 고치면 됩니다.
이 ligand의 atom index는
2356 부터 2379 까지입니다.
공간 좌표만이 기록되어있고, conectivity가 없기 때문에
3HMM_receptor.pdb 파일에서 CONECT로 검색해서 해당 ligand의 번호에 해당되는 부분을 잘라내고, 3HMM_855.pdb에 붙여 넣습니다.


남은 HETATM 는 물분자입니다. docking 준비 과정에서 물분자를 제거하는 옵션이 있긴 한데, 수작업으로 제거하되, 꼭 필요한 것만 남길 수도 있습니다.
pymol로 살펴봤을 때, residue idx 1006 의 물분자는 리간드와의 상호작용에서 중요한 역할을 하기에 남기고 나머지는 다 지워줍니다. 이렇게 설정할 경우, autodock_tools의 prepare_receptor4.py에서 물분자 제거 옵션을 사용하지 않아야 합니다.
ligand 파일은 다음과 같습니다.
다음은 chimera를 사용한 도킹입니다.
일단 UCSF chimera를 실행합니다.
file에서 open을 누릅니다. 사용할 파일을 선택합니다.

구조가 표시됩니다.

tools에서 surface/binding analysis -> Autodock Vina를 선택합니다. 우측과 같은 도킹 옵션 창이 나옵니다.
output file은 out.pdbqt 라고 만들어봅시다.
receptor와 ligand는 파일에 맞게 설정해줍니다.
docking box를 설정해야 하는데, 보통 box size는 22.5, 22.5, 22.5 정도를 사용하지만 구조를 보면서 가변적으로 바꿀 수 있습니다.
한 pdb 파일만이 아니라, 다양한 pdb 파일의 리간드들이 다 box 안에 들어오도록 잡는 것이 좋습니다.
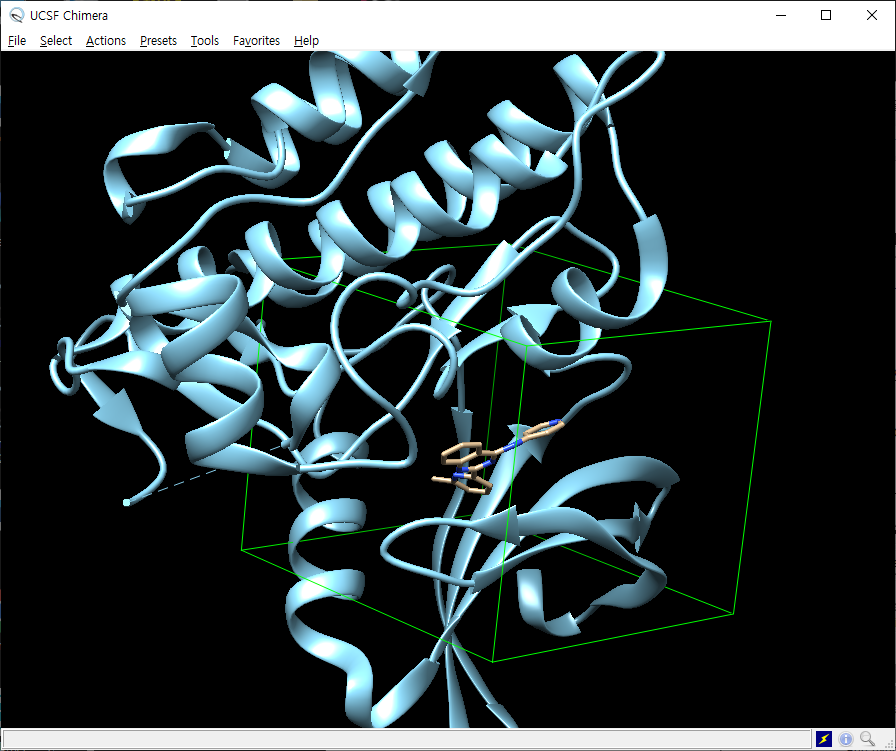

box 사이즈를 업데이트하면 화면에 박스가 표시됩니다.
receptor options나 ligand options는 autodock tools의 prepare_receptor4.py 와 prepare_ligand4.py를 실행하기 위한 옵션입니다. chimera 내부에도 python 과 autodock tools 등이 포함되어 있습니다. 다만 python 2 버전입니다. 따로 설치해둔 autodock tools와는 별개로 작동합니다. Ignore waters 만 false로 바꾸고 나머지는 기본값을 사용합니다.
number of binding modes는 최종적으로 출력될 binding modes의 수인데, 기본값은 9이지만 10으로 바꿨습니다. 별로 중요하진 않습니다.
executable location에는 다운로드한 vina 나 smina 의 실행파일 경로를 입력합니다.
OK 버튼을 누르면 도킹 시뮬레이션이 시작됩니다.
도킹이 끝나면 화면에 구조가 표시됩니다.
몇몇 경고가 나올 수도 있지만, 실행에는 지장이 없습니다.

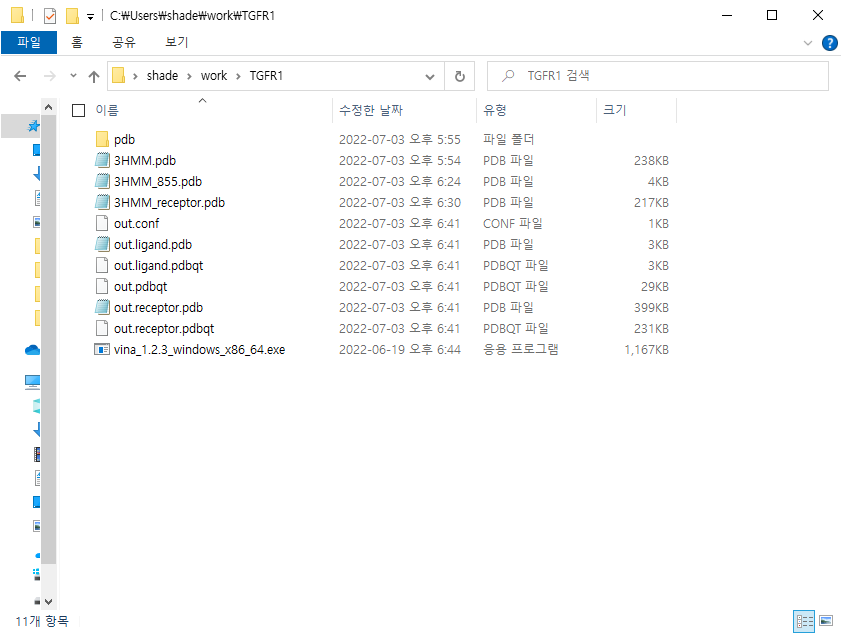
작업 경로에 out이라는 이름의 파일들이 생겼습니다.
out.conf 파일은 도킹 옵션(box size 등이 기록된 파일입니다. 텍스트로 열어볼 수 있습니다.
나중에 콘솔로 도킹을 실행할 때 vina --config out.conf 를 입력하면 이 파일에 기록된 옵션들을 인자로 받을 수도 있습니다.
out.receptor.pdb 는 3HMM_receptor.pdb 에서 몇 가지 fix가 진행된 파일입니다. 위에서 설정한 receptor options 이 반영되어 수소 원자가 추가되었습니다. out.receptor.pdbqt 는 이 파일을 docking에 필요한 pdbqt 파일로 변환된 것입니다.
out.ligand.pdb는 ligand options 이 적용된 파일로, 3D 좌표도 0,0,0 근방으로 바뀌어 있습니다. out.ligand.pdbqt는 이 파일을 docking용 확장자로 변환한 것입니다. vina 같은 경우 rotatable bond만을 고려하고 그 이외의 조각들은 강체로 취급하기에 bond length나 bond angle은 바뀌지 않고 torsional angle 만 바뀝니다. pdbqt는 bond 정보를 제외하고 강체 조각들의 연결로 구조를 표현한 확장자입니다.
out.pdbqt 파일이 도킹 결과 파일입니다.
MODEL 1부터 MODEL 10까지 도킹 결과 구조와 vina score 가 있습니다.
REMARK VINA RESULT: -10.554
이 파일은 pymol로도 열 수 있습니다.

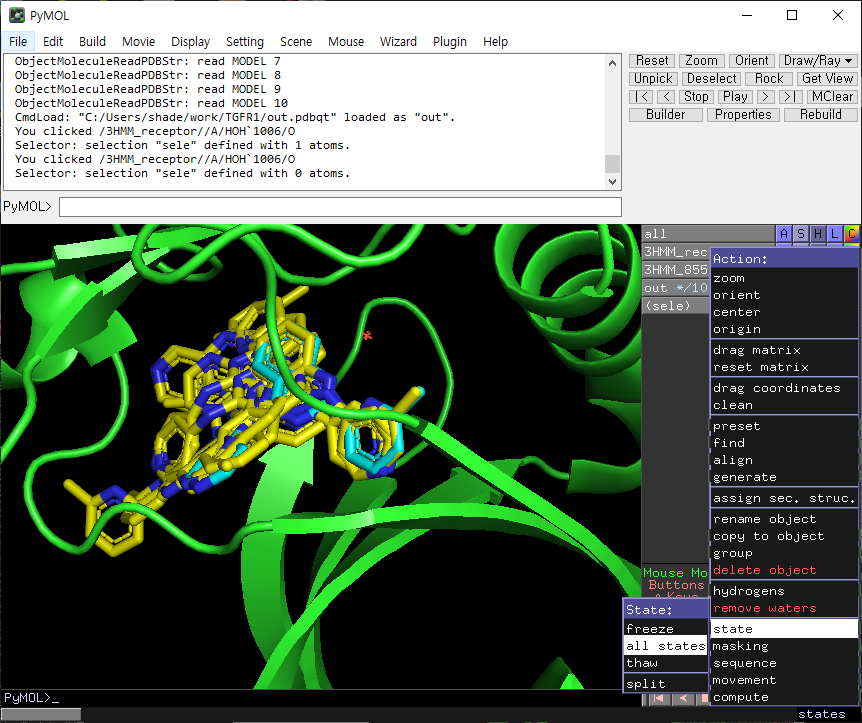
cyan이 crystal 구조, 노란색이 도킹된 구조입니다. 좌측은 model1만, 우측은 model 1부터 10 전체를 출력했습니다.
전체 출력은 A버튼(action)에서 state > all states 를 클릭하면 됩니다.
참고로 결과물 pdbqt 파일에
TORSDOF 3
ENDMDL
사이에 공백이 있는 경우가 있는데, 이럴 경우 pymol에서 구조를 제대로 읽지 못하고 model 1만 가져오는 버그가 있습니다. pymol 버전에 따라 문제없이 읽어지기도 합니다, 문제가 있을 경우 pdbqt파일에서 공백을 지우고 저장하시면 됩니다.
여기선 pdb에 붙어있던 ligand 파일을 초기 구조로 사용했지만, 새로운 ligand의 경우는 3D 구조를 만들어야 합니다.
openbabel 을 사용하면 분자의 SMILES로부터 3D 구조를 만들 수 있습니다.
일단 pdb파일로부터 3D 구조는 anaconda prompt 에서 다음과 같이 입력하면 만들 수 있습니다.
(base) C:\Users\shade\work\TGFR1>obabel 3HMM_855.pdb -osmi
Cc1cccc(n1)C1=[N]=C(c2ccccc2N1)Nc1ccncc1 3HMM_855.pdb
1 molecule converted
obabel은 openbabel을 설치하면 사용할 수 있습니다.
파일에 저장된 구조가 아니라 직접 그린 구조에서 SMILES를 얻고 싶다면
https://novelism.tistory.com/241
를 참고하세요.
SMILES로부터 3D 초기구조를 얻는 명령어는 다음과 같습니다.
(base) C:\Users\shade\work\TGFR1>obabel -:"Cc1cccc(n1)C1=[N]=C(c2ccccc2N1)Nc1ccncc1" --gen3d -O input.pdb
1 molecule converted
위의 예시대로 chimera에서 이 파일을 사용해서 도킹을 진행하면 됩니다.
'Drug > Computer-Aided Drug Discovery' 카테고리의 다른 글
| reference 를 이용하여 pdbqt를 pdb 로 변환하기 (0) | 2022.08.29 |
|---|---|
| 알파폴드가 예측한 단백질 구조는 완벽한가? (3) | 2022.08.05 |
| chimera GUI에서 docking (vina) 사용법: 2. pdb 파일 구하기 (0) | 2022.07.03 |
| chimera GUI에서 docking (vina) 사용법: 1. 프로그램 설치 (0) | 2022.07.03 |
| conda 로 pymol, openbabel 설치시 주의사항 (0) | 2022.06.26 |