딥러닝으로 분자의 다양한 특성을 예측하는 연구는 최근 수년 사이에 인기 있는 주제 중 하나입니다.
여러 특성들 중에서도 신약개발에서 가장 중요한 task는 분자의 bioactivity (단백질에 대한 결합, 혹은 효능)을 예측하는 것일 것입니다.
하지만, 다른 특성 예측과 비교하면 bioactivity 예측은 정확도가 낮습니다.
저는 그 이유가 단지 머신의 아키텍처나 학습 방식의 문제가 아니라고 생각합니다.
좀 더 근본적으로 데이터 자체가 가지는 문제를 생각해볼 수 있습니다. 호
머신러닝은 말 그대로 데이터로부터의 학습이고, 통계 이론의 적용을 받습니다.
통계에서 하는 일은, 표본 데이터로부터, 모집단의 확률분포를 유추하는 것이고, 머신러닝도 크게 다르지 않습니다.
그런데, 학습에 사용하는 데이터가 추정해야 할 집단의 데이터와 전혀 다른 분포를 가지고 있다면 어떨까요?
bioactivity 예측에서 많이 사용하는 데이터로 ChEMBL이 있습니다.
ChEMBL의 데이터를 잘 살펴보면, 데이터의 분포에서 특이한 점을 찾을 수 있습니다.
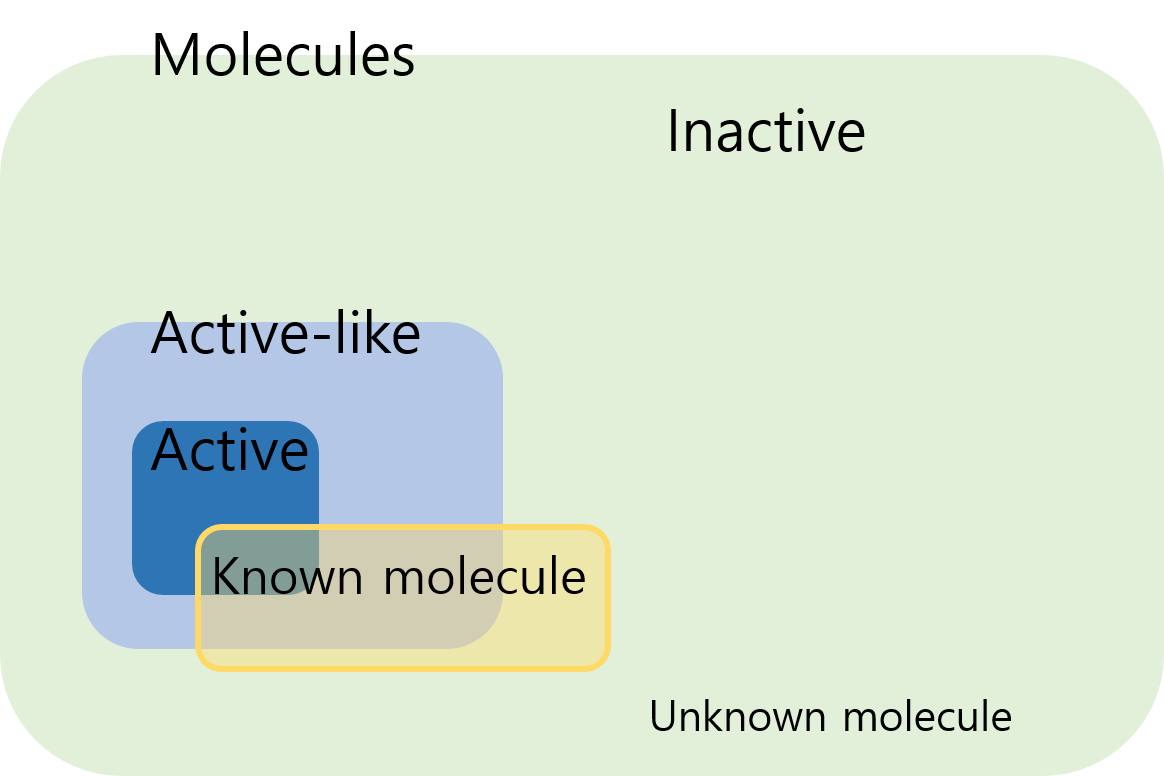
위의 그림은 하나의 예시입니다. 전체 분자들 중, 알려진 분자들(ChEMBL에 activity 정보가 있는 분자들)은 알려지지 않은 분자에 비해 극소수입니다. High-throughput screening 실험에서 보통 hit ratio는 1000~3000개 중 1개 정도 수준입니다. 그런데, ChEMBL에 수집된 분자들에선 active 비율이 상당히 높습니다. 경우에 따라 다르지만, IC 50을 기준으로 한다면, 50% 정도가 active입니다.
ChEMBL는 논문, 특허 등으로부터 수집된 데이터로 이루어져 있습니다. 여기서 생각해볼 점은, inactive 가 수집될 수 있는가 라는 것입니다. bioactive 분자 위주로 수집하는 DB에, 굳이 다양한 inactive들이 수집되기는 어렵습니다. 누가 inactive로 특허를 내고 논문을 쓰겠습니까?
그런데, inactive들이 있긴 있습니다. 이들의 정체는 무엇일까요? 논문에 들어있는 inactive분자들은 뭘까요...
ChEMBL을 살펴보면, 수~수십 개의 분자들의 레퍼런스가 특정 한 편의 논문인 경우가 많습니다. 이것은, lead optimize 과정에서 분자 유사체 (analog)에 대해 activity 실험을 하고 QSAR 분석을 하는 과정에서 나온 결과물들입니다. 대부분 동일한 코어 구조를 가지고 있고, R 그룹들을 변화시키면서 activity의 변화를 살펴본 것들입니다. 이것들은, 비록 inactive라 할지라도, 구조적으로는 active와 크게 다르지 않습니다. 저는 이들을 active-like라고 부르겠습니다.
데이터의 절대적인 수보다도, 데이터의 분포가 과도하게 편향되어 있다는 점이 문제입니다.
이런 데이터를 이용해서 activity를 학습하고, 학습 데이터와 전혀 분포가 다른 다양한 분자들에 대해서 activity를 예측하면 어떻게 될까요? 예측해야 될 대부분 분자는 unknown입니다. active와 매우 유사한 분자는 높은 confidence 값을, active-like와 유사한 분자에 대해선 매우 낮은 confidence값을, 그 외 분자에 대해선 아마도 중간값이 나올 것입니다. 이것이 우리가 원하는 예측 결과인가요? 만약 우리가 lead optimize를 하려 하는 것이고, 학습에 사용한 분자들이 특허를 보유한 분자들이라면 아마도 괜찮은 결과가 나올 것입니다. 그런데, hit compounds를 탐색하려는 것이라면 어떤가요? 이 경우 우리가 찾고 싶은 분자는 active이지만, 신규성을 가져야 하므로 이런 형태의 학습으로 적절한 결과를 얻기를 기대하기 어렵습니다.
이런 데이터의 편향 문제는, docking이나 pharmacophore 모델의 validation에서도 비슷하게 나타납니다. 알려진 데이터에 대한 예측 정확도는 높지만, 새로운 분자들에 대해 실험을 해보면 예측 정확도가 그보다 못한 경우들이 있습니다. 그래도 ML에 비하면 문제의 심각성이 적습니다.
저는 여기서 조금 다른 방법을 시도해봤습니다. 데이터셋에서 active가 아닌, active+active-like 분자를 선별해내는 것입니다. 그리고 active/active-like 를 분류하는것은 docking 스코어를 사용합니다.
semi-supervised learning 에 해당된다고 볼 수 있습니다.
active, active-like를 하나로 묶어서 active-like 라는 label 로 통합하고,
예측해야할 데이터셋을 inactive label로 취급하고 이를 10-fold로 split합니다.
이제, active-like와 inactive로 데이터를 학습할것입니다. 학습은 총 10번 할것인데, 매번 사용하는 active-like 데이터는 동일하게 고정하고, inactive 데이터만을 바꿔줍니다. 첫번째 학습에선 1st- fold를 택해서 데이터를 학습한 후, 이 모델로 나머지 9-fold에 대해서 예측합니다. 2nd fold를 택해서 데이터를 학습한 후, 이 모델로 나머지 9-fold에 대해서 예측합니다. 이과정을 10개의 fold에 대해서 반복합니다.
이러면 한 데이터당 9개의 예측 결과가 나올텐데, 이들의 평균값을 컨센서스 스코어로 사용합니다.
Random forest를 사용해서 학습한 결과, active-like와 inactive 판별이 눈으로 봤을 때 그럴듯하게 나오더군요.
다만 제가 시도한 타겟의 경우, 데이터의 양 자체가 적고 (50개) imbalance가 심해서 딥러닝(graph conv)로 학습하기 어려웠습니다. graph conv를 쓰고싶다면, 미리 대량의 데이터로 프리트레이닝하거나, 다른 방법을 활용해야할 것 같습니다. active-like라고 해서 전부 신규성이 부족하진 않습니다. core 구조 하나만 일치하고, 스케폴드 자체는 다른 경우들도 있습니다.
'Drug > Computer-Aided Drug Discovery' 카테고리의 다른 글
| 인공지능 신약개발에서 데이터 생산의 중요성 (0) | 2022.03.19 |
|---|---|
| Enrichment Factor (0) | 2022.03.13 |
| 인공지능 신약개발 무료 강의 (1) | 2022.02.13 |
| 인공지능 신약개발 분야의 현재와 짧은 미래의 이슈 (3) | 2022.02.09 |
| fragment 기반 약물 가상탐색 (0) | 2022.02.03 |