pymol에 다양한 기능이 있지만, 그중 많이 사용하는 기능 중 하나가 단백질-리간드 상호작용 보기입니다.
예시 PDB 는 EGFR kinase domain (wild type)에 Iressa (gefitinib)가 붙은 구조입니다.
pdb ID 는 2ITY이고, 리간드 ID는 IRE입니다.
다운로드하고 pymol을 실행합니다.
$ wget https://files.rcsb.org/download/2ITY.pdb
$ pymol 2ITY아래 같은 화면이 출력됩니다.

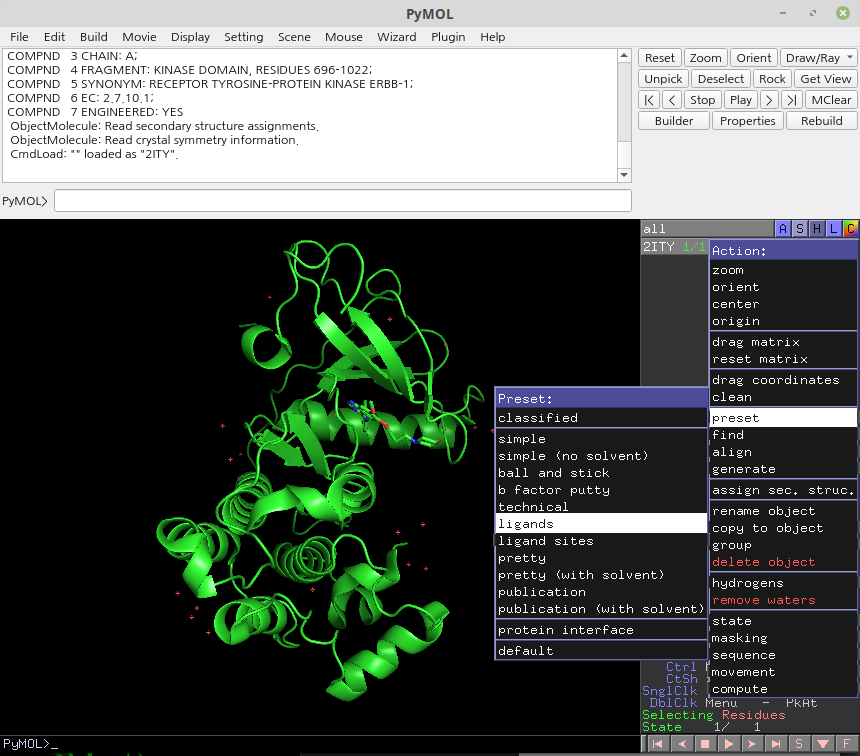
여기서 2ITY 옆의 A 버튼 (Action)을 누릅니다.
그러면 preset 설정들이 나오는데, 이중 ligands를 선택합니다.
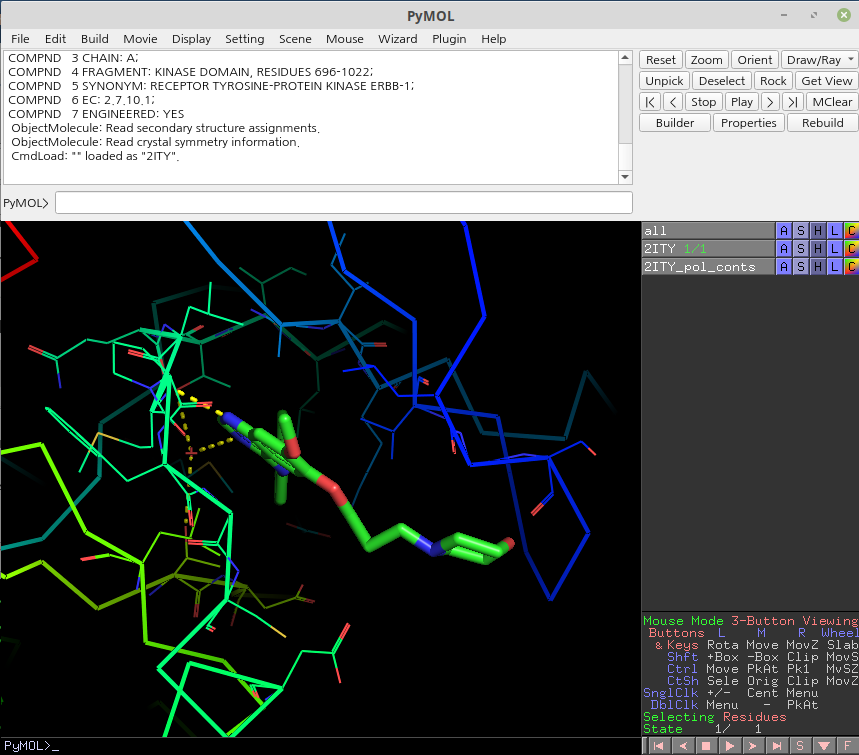
그럼 우측처럼 단백질 구조가 ribbon diagram으로 바뀌고, 리간드 중심으로 화면이 포커스 됩니다. ligand 주변 residue들이 lines로 표현됩니다. 상호작용이 있을 경우 노란 점선으로 표시해줍니다.
표시되는 상호작용은 수소결합 (water 포함), cation-anion 상호작용, aromatic 상호작용, aromatic-cation 상호작용 (되던가...?)입니다.
rcsb pdb 사이트에서도 3D 뷰에서 이런 상호작용을 보여주는데, pymol에서 보여주는 것과 다른 경우가 있습니다.
프로그램마다 상호작용을 추출하는 방식이 다르고, 상호작용의 cutoff를 다르게 잡아서 그렇습니다.
예를 들면, 수소결합 acceptor에 F 를 포함시키는 프로그램도 있지만, 아닌 것들도 있습니다. 그리고 ligand에서 aromatics ring이 2개 이상 붙어있는 경우 잘못 식별되는 경우도 있는데, 이럴 때도 결과가 달라질 수 있습니다. 그래서 저는 직접 따로 만들어서 쓰고 있습니다.
ribbon diagram이 싫고, cartoon 이 좋다면 preset -> ligand sites -> cartoon을 선택해봅니다.
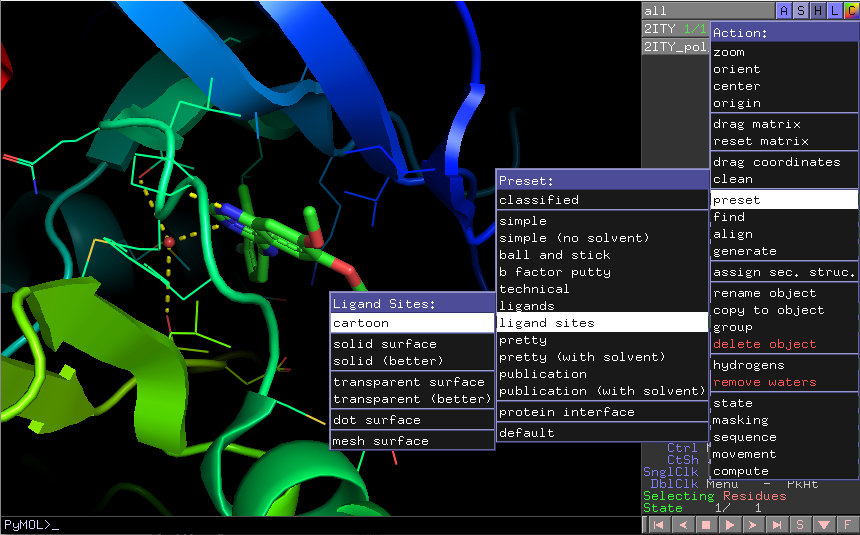

이외에도 다른 diagram들이 있으니 시도해보세요.
'Drug > Computer-Aided Drug Discovery' 카테고리의 다른 글
| PDB 파싱: CONECT 채워넣기 예제 (0) | 2022.01.29 |
|---|---|
| pymol 사용법: 구조 정렬 후 출력 (0) | 2022.01.24 |
| pymol 사용법: pymol open source 설치 (0) | 2022.01.24 |
| cheminformatics 툴킷: rd_filters (3) | 2022.01.18 |
| 인공지능 신약개발 분야에서 가장 기대되는 회사가 탄생했습니다. (0) | 2021.11.05 |