이전에 모종의 이유로 3d conformer를 만들 때 주로 openbabel을 사용했는데, 이제 rdkit으로 갈아타야겠습니다.
NCGC00257649
COc1ccc(CCN2CCC(CC2)Nc2nc3ccccc3n2Cc2ccc(F)cc2)cc1

이렇게 생긴 분자를 obabel과 rdkit으로 3D conformer를 생성해 봤습니다.
obabel -:"COc1ccc(CCN2CCC(CC2)Nc2nc3ccccc3n2Cc2ccc(F)cc2)cc1" -O g.pdb
보시는 것처럼 aromatic ring이 망가졌습니다.
aromatic bond나 double bond로 연결된 원자들은 한 평면에 있어야 합니다만... bond order가 잘못 인식되었는지 저렇게 되어버리네요.
SMILES를 다음처럼 Kekule form으로 바꿔도 마찬가지입니다.
COC1=CC=C(CCN2CCC(CC2)NC2=NC3=CC=CC=C3N2CC2=CC=C(F)C=C2)C=C1
from rdkit import Chem
from rdkit.Chem import AllChem
mol_name = 'NCGC00257649'
smi = 'COc1ccc(CCN2CCC(CC2)Nc2nc3ccccc3n2Cc2ccc(F)cc2)cc1'
m = Chem.MolFromSmiles(smi)
m2 = Chem.AddHs(m)
AllChem.EmbedMolecule(m2)
Chem.MolToMolFile(m2, mol_name +'.mol')NCGC00257649.pdb
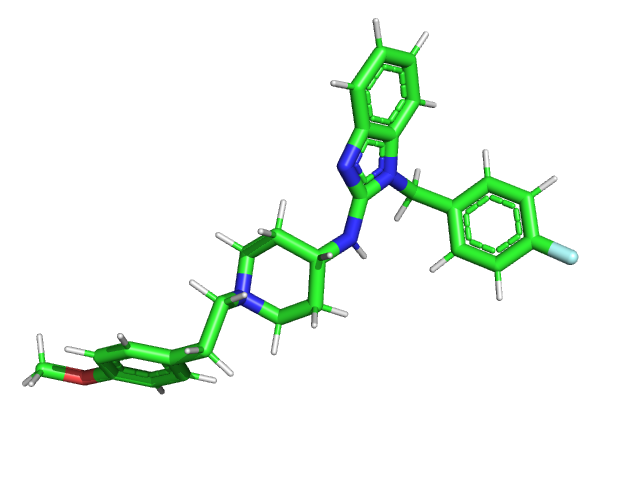
rdkit으로 생성한 3d conformer에선 5-membered ring이 정상적으로 한 평면상에 있습니다.
rdkit으로 생성이 제대로 안 되는 경우도 있을 수 있겠지만... 대규모로 사용해보지 않으면 모르겠네요.
'Drug > Computer-Aided Drug Discovery' 카테고리의 다른 글
| FragDockRL (6) | 2025.08.14 |
|---|---|
| SMINA를 이용한 tethered docking (2) | 2025.08.13 |
| hit와 lead 의 차이 - hit, hit to lead, lead optimazation (2) | 2023.06.17 |
| 인공지능 신약개발 회사 목록 (3) | 2022.12.16 |
| 단백질 general 서열생성 모델과 구조 예측 (3) | 2022.11.15 |